プレスリリース
複雑な形状を持つタンパク質をゼロから人工設計することに成功【自然科学研究機構(分子科学研究所/生命創成探究センター)、理化学研究所、大阪大学、千葉大学】
自然科学研究機構分子科学研究所協奏分子システム研究センター/生命創成探究センター生命分子創成研究グループの佐久間航也総合研究大学院大学大学院生(当時)、小林直也特任研究員(現:奈良先端科学技術大学院大学助教)、小杉貴洋助教、古賀(巽)理恵特任研究員(現:大阪大学蛋白質研究所助教)、古賀信康教授(現:大阪大学蛋白質研究所教授)は、理化学研究所生命機能科学研究センターの小林直宏上級研究員、長島敏雄上級テクニカルスタッフ、大阪大学蛋白質研究所の杉木俊彦特任助教(現:北里大学薬学部准教授)、藤原敏道教授(現:大阪大学名誉教授)、千葉大学大学院理学研究院の鈴木花野特任研究員(現:同特任助教)、村田武士教授と共同で、αヘリックスが不規則に並ぶ複雑な形状のall α型タンパク質の立体構造を人工設計する技術を開発し、これを用いることで、5本または6本のαヘリックスからなる5つの異なる複雑な形状のall α型タンパク質の人工設計に世界で初めて成功しました。本研究成果は、学術誌「Nature Structural and Molecular Biology」に2024年1月4日付でオンライン掲載されました。
【発表のポイント】
・これまで設計が困難であったαヘリックスが不規則に並ぶ複雑な形状のall α型タンパク質の人工設計に、世界で初めて成功した。
・シンプルな形状から複雑な形状まで多様な立体構造を生成できることを計算機シミュレーションで示し、生化学実験により実際に設計可能であることを示した。
・今回の研究で、多様で複雑な形状のタンパク質を設計することが可能になるため、機能性タンパク質の創出につながることが期待される。
研究グループは、タンパク質の2次構造(#1)がαヘリックスのみから構成されるall α型タンパク質について、自然界に典型的に見られる18個のヘリックス-ループ-ヘリックス(HLH)モチーフと、ヘリックスの長さを組み合わせることで、αヘリックスが平行に並ぶシンプルな形状から、不規則に並ぶ複雑な形状まで多様な立体構造を生成できることを計算機シミュレーションで示しました。そして、生成した構造の中から5つの異なる構造を選び、これらに折り畳むアミノ酸配列を計算機で人工設計し、生化学実験でそれらの折り畳み能を実証しました。これにより、計算機で生成した多様な立体構造が実際に設計可能であることを示しました。タンパク質の機能はその立体構造に基づいて発現されることから、多様で複雑な形状のタンパク質を設計することで、新規機能性タンパク質の創出につながることが期待されます。
1.研究の背景
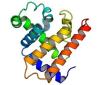
タンパク質はアミノ酸配列に従って特定の立体構造に折り畳み、その立体構造にもとづいて機能を発現します。タンパク質の立体構造の形状は、規則的な水素結合パターンからなるβシートおよびαヘリックスと呼ばれる2次構造の3次元的な配置で大まかに決まります。しかしながら、多くの自然界のタンパク質の立体構造は、これら2次構造の配置に対称性や規則性を見出すことが難しい複雑な形状をしており、世界で初めてタンパク質立体構造を決定したKendrewは、決定したミオグロビンの立体構造(図1)を見て "difficult to describe the arrangement in simple terms(そのヘリックスの配置を簡単な言葉で表現することは難しい)(J. C. Kendrew et al., 181, 662, Nature, 1958)"と述べています。
近年、タンパク質の立体構造を主鎖を含めてゼロから設計することで、自然界に存在するタンパク質とは異なる新しいタンパク質を生み出す技術が進展しています。しかしながら、2次構造がαヘリックスのみからなるall α型タンパク質については、これまでの人工設計のほとんどが、αヘリックスが構造中でほぼ平行に並ぶシンプルな形状をしており、自然界に見られるような複雑な形状を持つタンパク質を設計するための技術開発が求められていました。そこで研究チームは、不規則に並ぶαヘリックスからなる複雑な形状のall α型タンパク質を設計する技術の開発を試みました。
・図1:ミオグロビンの立体構造
リボンは2次構造であるαヘリックスを表す。構造を見やすくするため、ミオグロビンが結合するヘムは省略した。
2.研究の成果
研究グループは、まず自然界のタンパク質構造データベース(PDB: https://pdbj.org/?lang=ja
)を解析することで、自然界に典型的に観測され、異なる折れ曲がり角度を持つHLHモチーフを18個特定しました(図2)。次に、これら18個の典型的なHLHモチーフとヘリックスの長さの組み合わせを網羅的に変えながら、コンパクトな構造を持つタンパク質を計算機で生成したところ、αヘリックスが平行に並ぶシンプルな構造から、αヘリックスが不規則に並ぶ複雑な構造まで、多様なall α型タンパク質の主鎖構造を生成できることを見出しました(図3)。
続いて研究グループは、5本または6本のαヘリックスからなる生成した主鎖構造の中から、5つの異なる複雑な形状の構造を選択し、これらの形状に折り畳むようなアミノ酸配列(側鎖構造)を計算機で設計し、生化学実験で折り畳み能を検証しました。実験の結果、設計したタンパク質は単量体で折り畳み、変性温度は100℃以上の非常に安定な構造を形成していることが明らかになりました。さらに、これら設計したタンパク質の実験構造を核磁気共鳴分光法(#2)およびX線結晶構造解析法(#3)により決定したところ、設計通りの3次構造(#1)に折り畳んでいることが明らかになりました(図4)。
このようにして、研究グループは複雑な形状のall α型タンパク質の人工設計に成功しました。
・図2:18個の典型的なHLHモチーフ
自然界で典型的に観測されるHLHモチーフ18個それぞれが、リボンで表されたαヘリックスと、それらを連結するループで表されている。球はターンを構成する、ひとつずつの アミノ酸残基を表現し、球の色はそれらアミノ酸残基のおおよその局所的な形状を表す。折れ曲がり角度の程度によって、ヘアピン型(h)、Vシェイプ型(v)、コーナー型(c)の3つに分類されている。
・図3:18個の典型的なHLHモチーフを用いて計算機で生成した6本ヘリックス構造の複雑さの分布
hはヘアピン型のHLHモチーフのみを用いて構造を生成した場合、h+vはヘアピン型およびVシェイプ型のHLHモチーフを用いて構造を生成した場合、h+v+cは、コーナー型を含む全18個のHLHモチーフを用いて構造を生成した場合の結果が示されている。横軸の「構造の複雑さ」は、αヘリックスがお互いにどの程度平行に並んでいるかで定義され、数値が0に近づくほど構造が複雑であることを示す。Vシェイプ型やコーナー型のHLHモチーフを含めることで、複雑な構造が生成される。グラフの上側に、生成された構造の例が示されている。
・図4:人工設計された5つの異なる複雑な形状のall α型タンパク質
左が計算機構造、右が実験構造(H5_fold-0_ElsaはX線結晶構造解析法で、その他の構造は核磁気共鳴分光法で決定された)。
3.今後の展開・この研究の社会的意義
本研究により、αヘリックスがタンパク質立体構造中で不規則に配置された複雑な形状のall α型タンパク質を人工設計することが可能になりました。複雑な形状のタンパク質構造を新規に人工設計することが可能になれば、その3次構造中に機能部位を構築できる可能性が高まります。本研究成果は、生命現象の制御・設計、医療等に貢献する新規機能性タンパク質の創成につながることが期待されます。
4.用語解説
(#1) タンパク質の階層構造:1次、2次、3次構造
タンパク質は1次から4次の階層構造を持つ。1次構造はアミノ酸の配列、2次構造はアミノ酸残基の主鎖間の規則的な水素結合で形成されるαヘリックスとβシートと呼ばれる局所的な構造、3次構造は3次元的に配置された2次構造がループで連結された構造、4次構造は複数のタンパク質分子が集まって形成される構造である。
(#2) 核磁気共鳴分光法
タンパク質を磁場中に置きラジオ波を照射することで生じる核スピンの共鳴現象を観測することにより、タンパク質の分子構造を原子レベルで解析することができる。
(#3) X線結晶構造解析法
タンパク質分子が3次元的に規則正しく並んだ結晶にX線を照射し、その回折像から結晶中の電子密度分布を計算することにより、タンパク質分子の構造を原子レベルで解析することができる。
5.論文情報
・掲載誌:Nature Structural and Molecular Biology
・論文タイトル:"Design of complicated all-α protein structures"(複雑なall-αタンパク質構造の人工設計)
・著者:Koya Sakuma†, Naohiro Kobayashi†, Toshihiko Sugiki, Toshio Nagashima, Toshimichi Fujiwara, Kano Suzuki, Naoya Kobayashi, Takeshi Murata, Takahiro Kosugi, Rie Tatsumi-Koga, and Nobuyasu Koga*(†共同筆頭著者、*責任著者)
・掲載日:2024年1月4日19時(オンライン公開)
・DOI:10.1038/s41594-023-01147-9 ( https://www.nature.com/articles/s41594-023-01147-9
)
6.研究グループ
分子科学研究所/生命創成探究センター
理化学研究所
大阪大学蛋白質研究所
千葉大学
7.研究サポート
本研究は、日本医療研究開発機構(AMED)創薬等ライフサイエンス研究支援基盤事業(BINDS)創薬等ライフサイエンス研究のための相関構造解析プラットフォームによる支援と高度化(創薬等ライフサイエンス研究のための多階層構造生命科学解析技術の支援と高度化)(JP19am0101072); 疾病関連膜タンパク質の生産および構造解析支援(JP20am0101083)、理研NMR研究基盤、科研費(15H05592; 古賀信康、18H05420; 古賀信康、小杉貴洋、18H05425; 村田武士、18K06152; 小林直宏)、JST戦略的創造研究推進事業さきがけ(JPMJPR13AD; 古賀信康)、JST未来社会創造事業(JPMJMI17A2; 小林直宏)、科研費特別研究員(15J02427; 佐久間航也)、自然科学研究機構 岡崎共通研究施設 計算科学研究センター(21-IMS-C174, 20-IMS-C157, 19-IMS-C175, 18-IMS-C155, 17-IMS-C147, 16-IMS-C129, 15-IMS-C180)、分子科学研究所 機器センター、基礎生物学研究所 生物機能情報分析室、放射光実験施設フォトンファクトリー等の支援を受けて実施されました。
【リリース発信元】 大学プレスセンター https://www.u-presscenter.jp/